我們將使用輪廓分數(shù)和一些距離指標來執(zhí)行時間序列聚類實驗,并且進行可視化
讓我們看看下面的時間序列:

如果沿著y軸移動序列添加隨機噪聲,并隨機化這些序列,那么它們幾乎無法分辨,如下圖所示-現(xiàn)在很難將時間序列列分組為簇:

上面的圖表是使用以下腳本創(chuàng)建的:
# Import necessary libraries
import os
import pandas as pd
import numpy as np
# Import random module with an alias 'rand'
import random as rand
from scipy import signal
# Import the matplotlib library for plotting
import matplotlib.pyplot as plt
# Generate an array 'x' ranging from 0 to 5*pi with a step of 0.1
x = np.arange(0, 5*np.pi, 0.1)
# Generate square, sawtooth, sin, and cos waves based on 'x'
y_square = signal.square(np.pi * x)
y_sawtooth = signal.sawtooth(np.pi * x)
y_sin = np.sin(x)
y_cos = np.cos(x)
# Create a DataFrame 'df_waves' to store the waveforms
df_waves = pd.DataFrame([x, y_sawtooth, y_square, y_sin, y_cos]).transpose()
# Rename the columns of the DataFrame for clarity
df_waves = df_waves.rename(columns={0: 'time',
1: 'sawtooth',
2: 'square',
3: 'sin',
4: 'cos'})
# Plot the original waveforms against time
df_waves.plot(x='time', legend=False)
plt.show()
# Add noise to the waveforms and plot them again
for col in df_waves.columns:
if col != 'time':
for i in range(1, 10):
# Add noise to each waveform based on 'i' and a random value
df_waves['{}_{}'.format(col, i)] = df_waves[col].apply(lambda x: x + i + rand.random() * 0.25 * i)
# Plot the waveforms with added noise against time
df_waves.plot(x='time', legend=False)
plt.show()
現(xiàn)在我們需要確定聚類的基礎(chǔ)。這里有兩種方法:
把接近于一組的波形分組——較低歐幾里得距離的波形將聚在一起。
把看起來相似的波形分組——它們有相似的形狀,但歐幾里得距離可能不低
距離度量
一般來說,我們希望根據(jù)形狀對時間序列進行分組,對于這樣的聚類-可能希望使用距離度量,如相關(guān)性,這些度量或多或少與波形的線性移位無關(guān)。
讓我們看看上面定義的帶有噪聲的波形對之間的歐幾里得距離和相關(guān)性的熱圖:

可以看到歐幾里得距離對波形進行分組是很困難的,因為任何一組波形對的模式都是相似的。例如,除了對角線元素外,square & cos之間的相關(guān)形狀與square和square之間的相關(guān)形狀非常相似

所有的形狀都可以很容易地使用相關(guān)熱圖組合在一起——因為類似的波形具有非常高的相關(guān)性(sin-sin對),而像sin和cos這樣的波形幾乎沒有相關(guān)性。
輪廓分數(shù)
通過上面熱圖和分析,根據(jù)高相關(guān)性分配組看起來是一個好主意,但是我們?nèi)绾味x相關(guān)閾值呢?看起來像一個迭代過程,容易出現(xiàn)不準確和大量的人工工作。
在這種情況下,我們可以使用輪廓分數(shù)(Silhouette score),它為執(zhí)行的聚類分配一個分數(shù)。我們的目標是使輪廓分數(shù)最大化。
輪廓分數(shù)(Silhouette Score)是一種用于評估聚類質(zhì)量的指標,它可以幫助你確定數(shù)據(jù)點是否被正確地分配到它們的簇中。較高的輪廓分數(shù)表示簇內(nèi)數(shù)據(jù)點相互之間更加相似,而不同簇之間的數(shù)據(jù)點差異更大,這通常是良好的聚類結(jié)果。
輪廓分數(shù)的計算方法如下:
- 對于每個數(shù)據(jù)點 i,計算以下兩個值:- a(i):數(shù)據(jù)點 i 到同一簇中所有其他點的平均距離(簇內(nèi)平均距離)。- b(i):數(shù)據(jù)點 i 到與其不同簇中的所有簇的平均距離,取最小值(最近簇的平均距離)。
- 然后,計算每個數(shù)據(jù)點的輪廓系數(shù) s(i),它定義為:s(i) = frac{b(i) - a(i)}{max{a(i), b(i)}}
- 最后,計算整個數(shù)據(jù)集的輪廓分數(shù),它是所有數(shù)據(jù)點的輪廓系數(shù)的平均值:text{輪廓分數(shù)} = frac{1}{N} sum_{i=1}^{N} s(i)
其中,N 是數(shù)據(jù)點的總數(shù)。
輪廓分數(shù)的取值范圍在 -1 到 1 之間,具體含義如下:
- 輪廓分數(shù)接近1:表示簇內(nèi)數(shù)據(jù)點相似度高,不同簇之間的差異很大,是一個好的聚類結(jié)果。
- 輪廓分數(shù)接近0:表示數(shù)據(jù)點在簇內(nèi)的相似度與簇間的差異相當(dāng),可能是重疊的聚類或者不明顯的聚類。
- 輪廓分數(shù)接近-1:表示數(shù)據(jù)點更適合分配到其他簇,不同簇之間的差異相比簇內(nèi)差異更小,通常是一個糟糕的聚類結(jié)果。
一些重要的知識點:
在所有點上的高平均輪廓分數(shù)(接近1)表明簇的定義良好且明顯。
低或負的平均輪廓分數(shù)(接近-1)表明重疊或形成不良的集群。
0左右的分數(shù)表示該點位于兩個簇的邊界上。
聚類
現(xiàn)在讓我們嘗試對時間序列進行分組。我們已經(jīng)知道存在四種不同的波形,因此理想情況下應(yīng)該有四個簇。
歐氏距離
pca = decomposition.PCA(n_components=2)
pca.fit(df_man_dist_euc)
df_fc_cleaned_reduced_euc = pd.DataFrame(pca.transform(df_man_dist_euc).transpose(),
index = ['PC_1','PC_2'],
columns = df_man_dist_euc.transpose().columns)
index = 0
range_n_clusters = [2, 3, 4, 5, 6, 7, 8]
# Iterate over different cluster numbers
for n_clusters in range_n_clusters:
# Create a subplot with silhouette plot and cluster visualization
fig, (ax1, ax2) = plt.subplots(1, 2)
fig.set_size_inches(15, 7)
# Set the x and y axis limits for the silhouette plot
ax1.set_xlim([-0.1, 1])
ax1.set_ylim([0, len(df_man_dist_euc) + (n_clusters + 1) * 10])
# Initialize the KMeans clusterer with n_clusters and random seed
clusterer = KMeans(n_clusters=n_clusters, n_init="auto", random_state=10)
cluster_labels = clusterer.fit_predict(df_man_dist_euc)
# Calculate silhouette score for the current cluster configuration
silhouette_avg = silhouette_score(df_man_dist_euc, cluster_labels)
print("For n_clusters =", n_clusters, "The average silhouette_score is :", silhouette_avg)
sil_score_results.loc[index, ['number_of_clusters', 'Euclidean']] = [n_clusters, silhouette_avg]
index += 1
# Calculate silhouette values for each sample
sample_silhouette_values = silhouette_samples(df_man_dist_euc, cluster_labels)
y_lower = 10
# Plot the silhouette plot
for i in range(n_clusters):
# Aggregate silhouette scores for samples in the cluster and sort them
ith_cluster_silhouette_values = sample_silhouette_values[cluster_labels == i]
ith_cluster_silhouette_values.sort()
# Set the y_upper value for the silhouette plot
size_cluster_i = ith_cluster_silhouette_values.shape[0]
y_upper = y_lower + size_cluster_i
color = cm.nipy_spectral(float(i) / n_clusters)
# Fill silhouette plot for the current cluster
ax1.fill_betweenx(np.arange(y_lower, y_upper), 0, ith_cluster_silhouette_values, facecolor=color, edgecolor=color, alpha=0.7)
# Label the silhouette plot with cluster numbers
ax1.text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))
y_lower = y_upper + 10 # Update y_lower for the next plot
# Set labels and title for the silhouette plot
ax1.set_title("The silhouette plot for the various clusters.")
ax1.set_xlabel("The silhouette coefficient values")
ax1.set_ylabel("Cluster label")
# Add vertical line for the average silhouette score
ax1.axvline(x=silhouette_avg, color="red", linestyle="--")
ax1.set_yticks([]) # Clear the yaxis labels / ticks
ax1.set_xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
# Plot the actual clusters
colors = cm.nipy_spectral(cluster_labels.astype(float) / n_clusters)
ax2.scatter(df_fc_cleaned_reduced_euc.transpose().iloc[:, 0], df_fc_cleaned_reduced_euc.transpose().iloc[:, 1],
marker=".", s=30, lw=0, alpha=0.7, c=colors, edgecolor="k")
# Label the clusters and cluster centers
centers = clusterer.cluster_centers_
ax2.scatter(centers[:, 0], centers[:, 1], marker="o", c="white", alpha=1, s=200, edgecolor="k")
for i, c in enumerate(centers):
ax2.scatter(c[0], c[1], marker="$%d$" % i, alpha=1, s=50, edgecolor="k")
# Set labels and title for the cluster visualization
ax2.set_title("The visualization of the clustered data.")
ax2.set_xlabel("Feature space for the 1st feature")
ax2.set_ylabel("Feature space for the 2nd feature")
# Set the super title for the whole plot
plt.suptitle("Silhouette analysis for KMeans clustering on sample data with n_clusters = %d" % n_clusters,
fontsize=14, fontweight="bold")
plt.savefig('sil_score_eucl.png')
plt.show()







可以看到無論分成多少簇,數(shù)據(jù)都是混合的,并不能為任何數(shù)量的簇提供良好的輪廓分數(shù)。這與我們基于歐幾里得距離熱圖的初步評估的預(yù)期一致
相關(guān)性
pca = decomposition.PCA(n_components=2)
pca.fit(df_man_dist_corr)
df_fc_cleaned_reduced_corr = pd.DataFrame(pca.transform(df_man_dist_corr).transpose(),
index = ['PC_1','PC_2'],
columns = df_man_dist_corr.transpose().columns)
index=0
range_n_clusters = [2,3,4,5,6,7,8]
for n_clusters in range_n_clusters:
# Create a subplot with 1 row and 2 columns
fig, (ax1, ax2) = plt.subplots(1, 2)
fig.set_size_inches(15, 7)
# The 1st subplot is the silhouette plot
# The silhouette coefficient can range from -1, 1 but in this example all
# lie within [-0.1, 1]
ax1.set_xlim([-0.1, 1])
# The (n_clusters+1)*10 is for inserting blank space between silhouette
# plots of individual clusters, to demarcate them clearly.
ax1.set_ylim([0, len(df_man_dist_corr) + (n_clusters + 1) * 10])
# Initialize the clusterer with n_clusters value and a random generator
# seed of 10 for reproducibility.
clusterer = KMeans(n_clusters=n_clusters, n_init="auto", random_state=10)
cluster_labels = clusterer.fit_predict(df_man_dist_corr)
# The silhouette_score gives the average value for all the samples.
# This gives a perspective into the density and separation of the formed
# clusters
silhouette_avg = silhouette_score(df_man_dist_corr, cluster_labels)
print(
"For n_clusters =",
n_clusters,
"The average silhouette_score is :",
silhouette_avg,
)
sil_score_results.loc[index,['number_of_clusters','corrlidean']] = [n_clusters,silhouette_avg]
index=index+1
sample_silhouette_values = silhouette_samples(df_man_dist_corr, cluster_labels)
y_lower = 10
for i in range(n_clusters):
# Aggregate the silhouette scores for samples belonging to
# cluster i, and sort them
ith_cluster_silhouette_values = sample_silhouette_values[cluster_labels == i]
ith_cluster_silhouette_values.sort()
size_cluster_i = ith_cluster_silhouette_values.shape[0]
y_upper = y_lower + size_cluster_i
color = cm.nipy_spectral(float(i) / n_clusters)
ax1.fill_betweenx(
np.arange(y_lower, y_upper),
0,
ith_cluster_silhouette_values,
facecolor=color,
edgecolor=color,
alpha=0.7,
)
# Label the silhouette plots with their cluster numbers at the middle
ax1.text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))
# Compute the new y_lower for next plot
y_lower = y_upper + 10 # 10 for the 0 samples
ax1.set_title("The silhouette plot for the various clusters.")
ax1.set_xlabel("The silhouette coefficient values")
ax1.set_ylabel("Cluster label")
# The vertical line for average silhouette score of all the values
ax1.axvline(x=silhouette_avg, color="red", linestyle="--")
ax1.set_yticks([]) # Clear the yaxis labels / ticks
ax1.set_xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
# 2nd Plot showing the actual clusters formed
colors = cm.nipy_spectral(cluster_labels.astype(float) / n_clusters)
ax2.scatter(
df_fc_cleaned_reduced_corr.transpose().iloc[:, 0],
df_fc_cleaned_reduced_corr.transpose().iloc[:, 1], marker=".", s=30, lw=0, alpha=0.7, c=colors, edgecolor="k"
)
# for i in range(len(df_fc_cleaned_cleaned_reduced.transpose().iloc[:, 0])):
# ax2.annotate(list(df_fc_cleaned_cleaned_reduced.transpose().index)[i],
# (df_fc_cleaned_cleaned_reduced.transpose().iloc[:, 0][i],
# df_fc_cleaned_cleaned_reduced.transpose().iloc[:, 1][i] + 0.2))
# Labeling the clusters
centers = clusterer.cluster_centers_
# Draw white circles at cluster centers
ax2.scatter(
centers[:, 0],
centers[:, 1],
marker="o",
c="white",
alpha=1,
s=200,
edgecolor="k",
)
for i, c in enumerate(centers):
ax2.scatter(c[0], c[1], marker="$%d$" % i, alpha=1, s=50, edgecolor="k")
ax2.set_title("The visualization of the clustered data.")
ax2.set_xlabel("Feature space for the 1st feature")
ax2.set_ylabel("Feature space for the 2nd feature")
plt.suptitle(
"Silhouette analysis for KMeans clustering on sample data with n_clusters = %d"
% n_clusters,
fontsize=14,
fontweight="bold",
)
plt.show()







當(dāng)選擇的簇數(shù)為4時,我們可以清楚地看到分離的簇,其他結(jié)果通常比歐氏距離要好得多。
歐幾里得距離與相關(guān)廓形評分的比較

輪廓分數(shù)表明基于相關(guān)性的距離矩陣在簇數(shù)為4時效果最好,而在歐氏距離的情況下效果就不那么明顯了結(jié)論
總結(jié)
在本文中,我們研究了如何使用歐幾里得距離和相關(guān)度量執(zhí)行時間序列聚類,并觀察了這兩種情況下的結(jié)果如何變化。如果我們在評估聚類時結(jié)合Silhouette,我們可以使聚類步驟更加客觀,因為它提供了一種很好的直觀方式來查看聚類的分離情況。
-
for循環(huán)
+關(guān)注
關(guān)注
0文章
61瀏覽量
2697
發(fā)布評論請先 登錄
【「時間序列與機器學(xué)習(xí)」閱讀體驗】全書概覽與時間序列概述
【《時間序列與機器學(xué)習(xí)》閱讀體驗】+ 了解時間序列
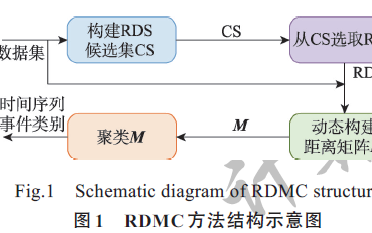
流式時間序列的實時相似度研究
基于u-shapelets的時間序列聚類算法
基于SAX的時間序列分類
基于層次劃分的密度優(yōu)化聚類算法
基于連續(xù)小波變換及其逆變換的聚類方法
基于動態(tài)時間彎曲距離的長期直覺模糊時間序列預(yù)測

如何使用無監(jiān)督形狀對時間序列進行聚類的資料說明

一種面向私有二進制協(xié)議的報文聚類方法





 使用輪廓分數(shù)提升時間序列聚類的表現(xiàn)
使用輪廓分數(shù)提升時間序列聚類的表現(xiàn)











評論